在上文拿到过滤的bam文件后,我们就可以进行接下来的质控了。
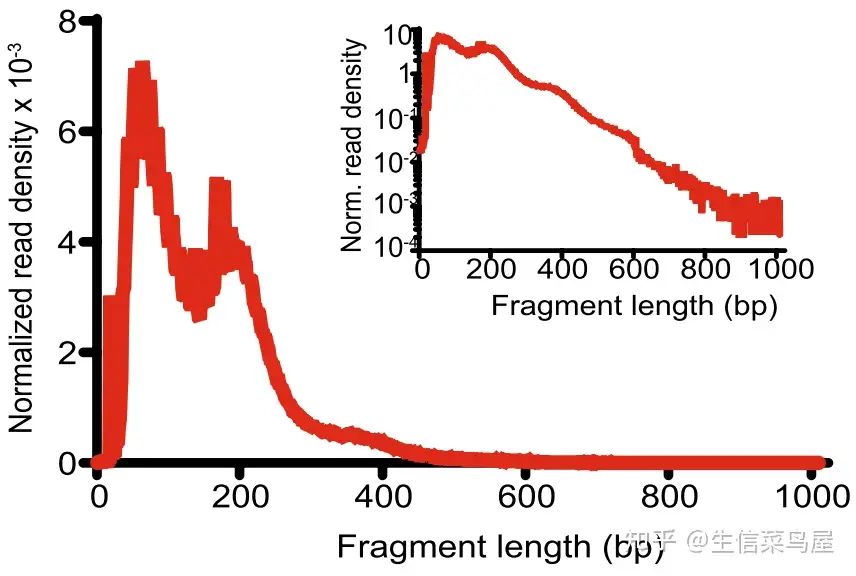
首先,ATAC-seq有自己独特的特征:1.ATAC-seq的片段分布是随着片段的增加而减少,在200bp,400bp的时候会有小峰出现。2.在TSS附近有明显的富集。
好了,现在我们来进行第一个质控。如何画ATAC-seq的fragment的分布呢?
samtools view -F 0x04 bam | awk -F'\t' 'function abs(x){return ((x < 0.0) ? -x : x)} {print abs($9)}' | sort | uniq -c | awk -v OFS="\t" '{print $2, $1/2}' >$projPath/alignment/sam/fragmentLen/${histName}_fragmentLen.txt #####提取片段长度
接下来使用R语言画图:
ggplot({histName}_fragmentLen,aes(x = fragLen, y = fragCount))+geom_line(size = 1)
理想情况下的片段分布如下图:
之后我们进行第二个质控,就是看TSS附近的富集程度:
bamCoverage --bam a.bam -o a.bw --normalizeUsing RPKM ####把bam转化为bigwig文件
computeMatrix reference-point --referencePoint TSS -p 15 -b 3000 -a 3000 -R gene.bed -S a.bw --skipZeros -o matrix_test_TSS.gz --outFileSortedRegions regions_test_genes.bed ####计算矩阵
plotHeatap -m matrix.mat.gz -out ExampleHeatmap1.png ###画图
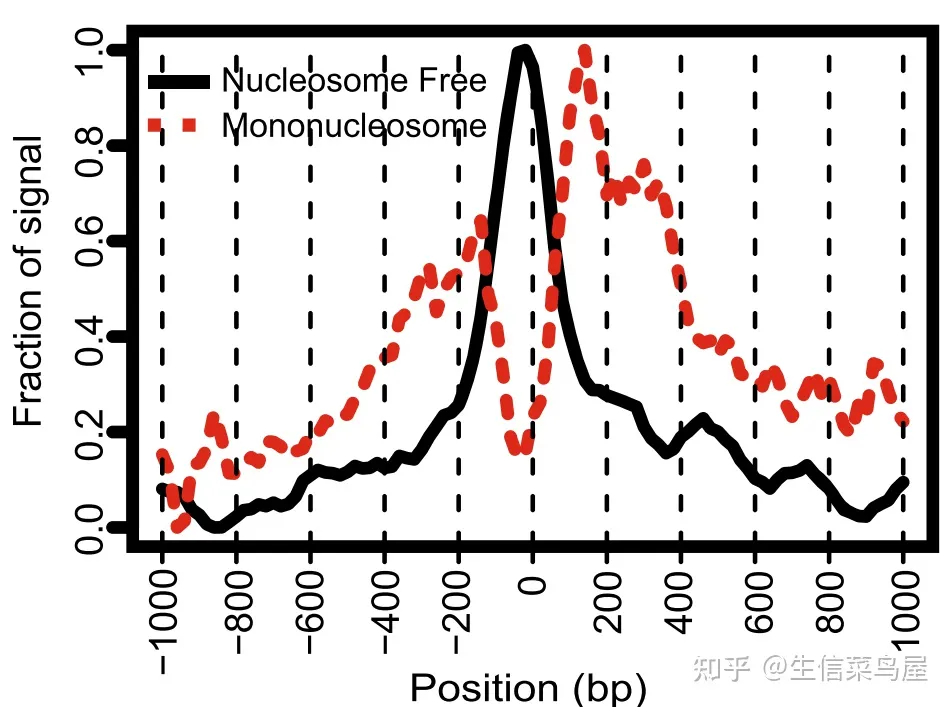
理想情况下的TSS富集图:
上面介绍了手动计算fragment以及TSS富集。有一个R包可以帮助我们直接完成上面的QC。这个工具包就是ATACseqQC,虽然使用方便;但是个人不太喜欢,因为速度实在是太慢了。有兴趣的同学可以去试试。 | ![]() 测序
255 人阅读
|
0 人回复
|
2024-07-22
测序
255 人阅读
|
0 人回复
|
2024-07-22