snpEff软件下载链接
https://sourceforge.net/projects/snpeff/files/
我下载的是 snpEff_v4_3t_core.zip
软件主页
http://snpeff.sourceforge.net/SnpEff_manual.html
帮助文档
http://snpeff.sourceforge.net/protocol.html
(软件不用安装解压出来就能够使用)
用大肠杆菌的一个数据做练习
在 snpEff.jar 同级目录下新建一个data文件夹
mkdir data
cd data
mkdir genomes
mkdir ecoli
大肠杆菌基因组和基因注释文件的下载链接
ftp://ftp.ncbi.nlm.nih.gov/genomes/all/GCF/000/005/845/GCF_000005845.2_ASM584v2/
下载基因组fasta和基因注释文件gff
基因组文件命名为ecoli.fa放在 genomes文件夹下
基因注释文件命名为genes.gff放到ecoli文件夹下
回到 snpEff.jar 同级目录 ,运行命令
echo "ecoli.genome:ecoli" >> snpEff.config
将 ecoli.genome:ecoli添加到 snpEff.config文件的最后一行
构建数据库
java -Xmx4G -jar snpEff.jar build -gff3 ecoli
在 data/ecoli目录下会多出一个 snpEffectPredictor.bin文件
对vcf文件进行注释
java -Xmx4G -jar snpEff.jar ecoli vcf_ann_Practice/E_coli_K12.filter.vcf > E_coli_K12.filter.eff.vcf
同时会生成一个 snpEff_summary.html 文件
在新生成的vcf文件里INFO列里会多出来很多信息
vcf文件不是很大的话可以用R语言的vcfR包对信息进行解析
读取vcf文件
annoV<-read.vcfR("E_coli_K12.filter.eff.vcf")
提取注释的信息
vcfR2tidy(annoV)$fix$ANN
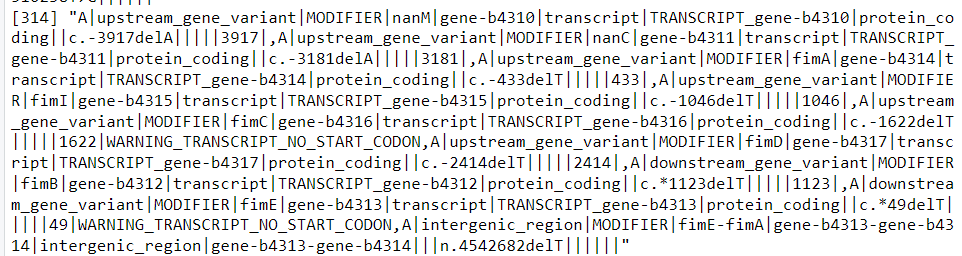
这里的内容很多,具体哪些信息有用暂时还没搞清楚
欢迎大家关注我的公众号
小明的数据分析笔记本
小明的数据分析笔记本 公众号 主要分享:1、R语言和python做数据分析和数据可视化的简单小例子;2、园艺植物相关转录组学、基因组学、群体遗传学文献阅读笔记;3、生物信息学入门学习资料及自己的学习笔记!