动动发财的小手,点个赞吧!

除了在网上找到的一些过度清理的数据集之外,缺失值无处不在。事实上,数据集越复杂、越大,出现缺失值的可能性就越大。缺失值是统计研究的一个令人着迷的领域,但在实践中它们往往很麻烦。
如果您处理一个预测问题,想要从 p 维协变量 X=(X_1,…,X_p) 预测变量 Y,并且面临 X 中的缺失值,那么基于树的方法有一个有趣的解决方案。这种方法实际上相当古老,但在各种数据集中似乎都表现得非常好。我说的是“缺失的属性标准”(MIA;[1])。虽然有很多关于缺失值的好文章(例如这篇文章),但这种强大的方法似乎有些未得到充分利用。特别是,不需要以任何方式插补、删除或预测缺失值,而是可以像完全观察到的数据一样运行预测。
我将快速解释该方法本身是如何工作的,然后提供一个示例以及此处解释的分布式随机森林 (DRF)。我选择 DRF 是因为它是随机森林的一个非常通用的版本(特别是,它也可以用来预测随机向量 Y),而且因为我在这里有些偏见。 MIA实际上是针对广义随机森林(GRF)实现的,它涵盖了广泛的森林实现。特别地,由于DRF在CRAN上的实现是基于GRF的,因此稍作修改后,也可以使用MIA方法。
当然,请注意,这是一个快速修复(据我所知)没有理论上的保证。根据缺失机制,分析可能会严重偏差。另一方面,处理缺失值的最常用方法没有任何理论保证,或者众所周知会使分析产生偏差,并且至少从经验上来看,MIA 似乎运作良好,并且
工作原理
回想一下,在 RF 中,分割的构建形式为 X_j < S 或 X_j ≥ S,维度 j=1,…,p。为了找到这个分割值 S,它优化了 Y 上的某种标准,例如 CART 标准。因此,观察结果通过依赖于 X 的决策规则连续划分。
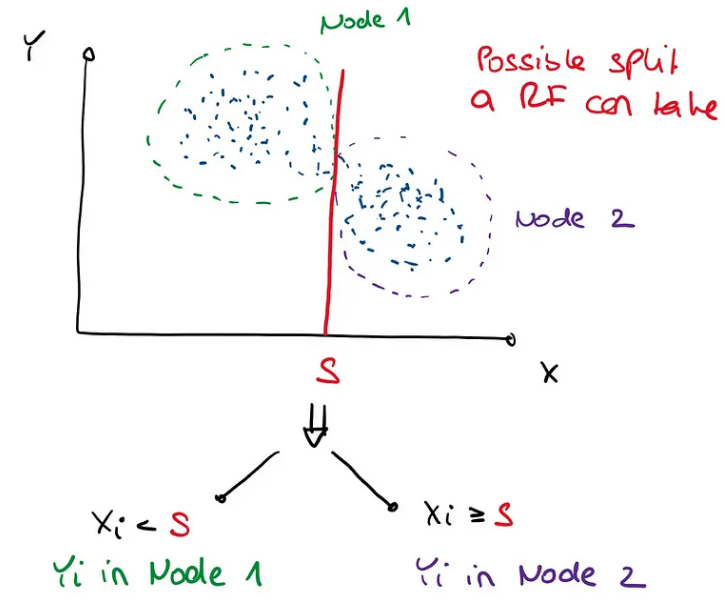
原论文的解释有点令人困惑,但据我了解,MIA 的工作原理如下:让我们考虑一个样本 (Y_1, X_1),…, (Y_n, X_n),
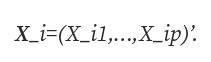
不缺失值的分割就是像上面那样寻找值S,然后将节点1中所有X_ij < S的Y_i和节点2中所有X_ij ≥ S的Y_i扔进去。计算每个值S的目标标准,例如CART,我们可以选择最好的一个。对于缺失值,每个候选分割值 S 有 3 个选项需要考虑:
- 对所有观测值 i 使用通常的规则,使得 X_ij 被观测到,如果 X_ij 丢失,则将 i 发送到节点 1。
- 对所有观测值 i 使用通常的规则,以便观测到 X_ij,如果缺少 X_ij,则将 i 发送到节点 2。
- 忽略通常的规则,如果 X_ij 缺失,则将 i 发送到节点 1;如果观察到 X_ij,则将 i 发送到节点 2。
遵循这些规则中的哪一个再次根据我们使用的 Y_i 的标准来决定。
例子
需要指出的是,CRAN 上的 drf 包尚未使用最新的方法进行更新。将来有一天,所有这些都将在 CRAN 上的一个包中实现。但是,目前有两个版本:
如果您想使用缺失值(无置信区间)的快速 drf 实现,您可以使用本文末尾附带的“drfown”函数。此代码改编自
lorismichel/drf: Distributional Random Forests (Cevid et al., 2020) (github.com)
另一方面,如果您想要参数的置信区间,请使用此(较慢的)代码
drfinference/drf-foo.R at main · JeffNaef/drfinference (github.com)
特别是,drf-foo.R 包含后一种情况所需的所有内容。
我们将重点关注具有置信区间的较慢代码,如本文所述,并考虑与所述文章中相同的示例:
set.seed(2)
n<-2000
beta1<-1
beta2<--1.8
# Model Simulation
X<-mvrnorm(n = n, mu=c(0,0), Sigma=matrix(c(1,0.7,0.7,1), nrow=2,ncol=2))
u<-rnorm(n=n, sd = sqrt(exp(X[,1])))
Y<- matrix(beta1*X[,1] + beta2*X[,2] + u, ncol=1)
请注意,这是一个异方差线性模型,p=2,误差项的方差取决于 X_1 值。现在我们还以随机缺失 (MAR) 方式向 X_1 添加缺失值:
prob_na <- 0.3
X[, 1] <- ifelse(X[, 2] <= -0.2 & runif(n) < prob_na, NA, X[, 1])
这意味着每当 X_2 的值小于 -0.2 时,X_1 缺失的概率为 0.3。因此X_1丢失的概率取决于X_2,这就是所谓的“随机丢失”。这已经是一个复杂的情况,通过查看缺失值的模式可以获得信息。也就是说,缺失不是“随机完全缺失(MCAR)”,因为X_1的缺失取决于X_2的值。这反过来意味着我们得出的 X_2 的分布是不同的,取决于 X_1 是否缺失。这尤其意味着删除具有缺失值的行可能会严重影响分析。
我们现在修复 x 并估计给定 X=x 的条件期望和方差,与上一篇文章中完全相同。
# Choose an x that is not too far out
x<-matrix(c(1,1),ncol=2)
# Choose alpha for CIs
alpha<-0.05
然后我们还拟合 DRF 并预测测试点 x 的权重(对应于预测 Y|X=x 的条件分布):
## Fit the new DRF framework
drf_fit <- drfCI(X=X, Y=Y, min.node.size = 5, splitting.rule='FourierMMD', num.features=10, B=100)
## predict weights
DRF = predictdrf(drf_fit, x=x)
weights <- DRF$weights[1,]
条件期望
我们首先估计 Y|X=x 的条件期望。
# Estimate the conditional expectation at x:
condexpest<- sum(weights*Y)
# Use the distribution of weights, see below
distofcondexpest<-unlist(lapply(DRF$weightsb, function(wb) sum(wb[1,]*Y) ))
# Can either use the above directly to build confidence interval, or can use the normal approximation.
# We will use the latter
varest<-var(distofcondexpest-condexpest)
# build 95%-CI
lower<-condexpest - qnorm(1-alpha/2)*sqrt(varest)
upper<-condexpest + qnorm(1-alpha/2)*sqrt(varest)
round(c(lower, condexpest, upper),2)
# without NAs: (-1.00, -0.69 -0.37)
# with NAs: (-1.15, -0.67, -0.19)
值得注意的是,使用 NA 获得的值与上一篇文章中未使用 NA 的第一次分析得到的值非常接近!这确实令我震惊,因为这个缺失的机制并不容易处理。有趣的是,估计器的估计方差也翻倍,从没有缺失值的大约 0.025 到有缺失值的大约 0.06。
真相如下:
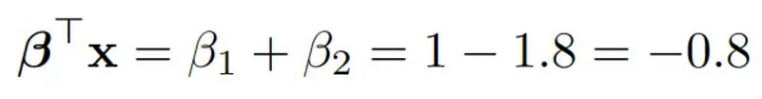
所以我们有一个轻微的错误,但置信区间包含事实,正如它们应该的那样。
对于更复杂的目标,结果看起来相似,例如条件方差:
# Estimate the conditional expectation at x:
condvarest<- sum(weights*Y^2) - condexpest^2
distofcondvarest<-unlist(lapply(DRF$weightsb, function(wb) {
sum(wb[1,]*Y^2) - sum(wb[1,]*Y)^2
} ))
# Can either use the above directly to build confidence interval, or can use the normal approximation.
# We will use the latter
varest<-var(distofcondvarest-condvarest)
# build 95%-CI
lower<-condvarest - qnorm(1-alpha/2)*sqrt(varest)
upper<-condvarest + qnorm(1-alpha/2)*sqrt(varest)
c(lower, condvarest, upper)
# without NAs: (1.89, 2.65, 3.42)
# with NAs: (1.79, 2.74, 3.69)
这里估计值的差异有点大。由于真相被给出为
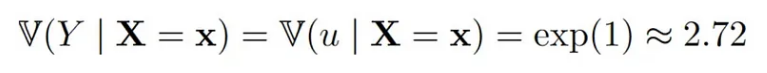
NA 的估计甚至稍微更准确(当然这可能只是随机性)。同样,(方差)估计量的方差估计随着缺失值的增加而增加,从 0.15(无缺失值)增加到 0.23。
结论
在本文中,我们讨论了 MIA,它是随机森林中分裂方法的一种改进,用于处理缺失值。由于它是在 GRF 和 DRF 中实现的,因此它可以被广泛使用,我们看到的小例子表明它工作得非常好。
然而,我想再次指出,即使对于大量数据点,也没有一致性或置信区间有意义的理论保证。缺失值的原因有很多,必须非常小心,不要因粗心处理这一问题而使分析产生偏差。 MIA 方法对于这个问题来说决不是一个很好理解的解决方案。然而,目前这似乎是一个合理的快速解决方案,它似乎能够利用数据缺失的模式。如果有人进行了更广泛的模拟分析,我会对结果感到好奇。
Code
require(drf)
drfown <- function(X, Y,
num.trees = 500,
splitting.rule = "FourierMMD",
num.features = 10,
bandwidth = NULL,
response.scaling = TRUE,
node.scaling = FALSE,
sample.weights = NULL,
sample.fraction = 0.5,
mtry = min(ceiling(sqrt(ncol(X)) + 20), ncol(X)),
min.node.size = 15,
honesty = TRUE,
honesty.fraction = 0.5,
honesty.prune.leaves = TRUE,
alpha = 0.05,
imbalance.penalty = 0,
compute.oob.predictions = TRUE,
num.threads = NULL,
seed = stats::runif(1, 0, .Machine$integer.max),
compute.variable.importance = FALSE) {
# initial checks for X and Y
if (is.data.frame(X)) {
if (is.null(names(X))) {
stop("the regressor should be named if provided under data.frame format.")
}
if (any(apply(X, 2, class) %in% c("factor", "character"))) {
any.factor.or.character <- TRUE
X.mat <- as.matrix(fastDummies::dummy_cols(X, remove_selected_columns = TRUE))
} else {
any.factor.or.character <- FALSE
X.mat <- as.matrix(X)
}
mat.col.names.df <- names(X)
mat.col.names <- colnames(X.mat)
} else {
X.mat <- X
mat.col.names <- NULL
mat.col.names.df <- NULL
any.factor.or.character <- FALSE
}
if (is.data.frame(Y)) {
if (any(apply(Y, 2, class) %in% c("factor", "character"))) {
stop("Y should only contain numeric variables.")
}
Y <- as.matrix(Y)
}
if (is.vector(Y)) {
Y <- matrix(Y,ncol=1)
}
#validate_X(X.mat)
if (inherits(X, "Matrix") && !(inherits(X, "dgCMatrix"))) {
stop("Currently only sparse data of class 'dgCMatrix' is supported.")
}
drf:::validate_sample_weights(sample.weights, X.mat)
#Y <- validate_observations(Y, X)
# set legacy GRF parameters
clusters <- vector(mode = "numeric", length = 0)
samples.per.cluster <- 0
equalize.cluster.weights <- FALSE
ci.group.size <- 1
num.threads <- drf:::validate_num_threads(num.threads)
all.tunable.params <- c("sample.fraction", "mtry", "min.node.size", "honesty.fraction",
"honesty.prune.leaves", "alpha", "imbalance.penalty")
# should we scale or not the data
if (response.scaling) {
Y.transformed <- scale(Y)
} else {
Y.transformed <- Y
}
data <- drf:::create_data_matrices(X.mat, outcome = Y.transformed, sample.weights = sample.weights)
# bandwidth using median heuristic by default
if (is.null(bandwidth)) {
bandwidth <- drf:::medianHeuristic(Y.transformed)
}
args <- list(num.trees = num.trees,
clusters = clusters,
samples.per.cluster = samples.per.cluster,
sample.fraction = sample.fraction,
mtry = mtry,
min.node.size = min.node.size,
honesty = honesty,
honesty.fraction = honesty.fraction,
honesty.prune.leaves = honesty.prune.leaves,
alpha = alpha,
imbalance.penalty = imbalance.penalty,
ci.group.size = ci.group.size,
compute.oob.predictions = compute.oob.predictions,
num.threads = num.threads,
seed = seed,
num_features = num.features,
bandwidth = bandwidth,
node_scaling = ifelse(node.scaling, 1, 0))
if (splitting.rule == "CART") {
##forest <- do.call(gini_train, c(data, args))
forest <- drf:::do.call.rcpp(drf:::gini_train, c(data, args))
##forest <- do.call(gini_train, c(data, args))
} else if (splitting.rule == "FourierMMD") {
forest <- drf:::do.call.rcpp(drf:::fourier_train, c(data, args))
} else {
stop("splitting rule not available.")
}
class(forest) <- c("drf")
forest[["ci.group.size"]] <- ci.group.size
forest[["X.orig"]] <- X.mat
forest[["is.df.X"]] <- is.data.frame(X)
forest[["Y.orig"]] <- Y
forest[["sample.weights"]] <- sample.weights
forest[["clusters"]] <- clusters
forest[["equalize.cluster.weights"]] <- equalize.cluster.weights
forest[["tunable.params"]] <- args[all.tunable.params]
forest[["mat.col.names"]] <- mat.col.names
forest[["mat.col.names.df"]] <- mat.col.names.df
forest[["any.factor.or.character"]] <- any.factor.or.character
if (compute.variable.importance) {
forest[['variable.importance']] <- variableImportance(forest, h = bandwidth)
}
forest
}