
 」
」
本期教程

本期教程,代码和数据可在社群中获得
往期教程部分内容













写在前面
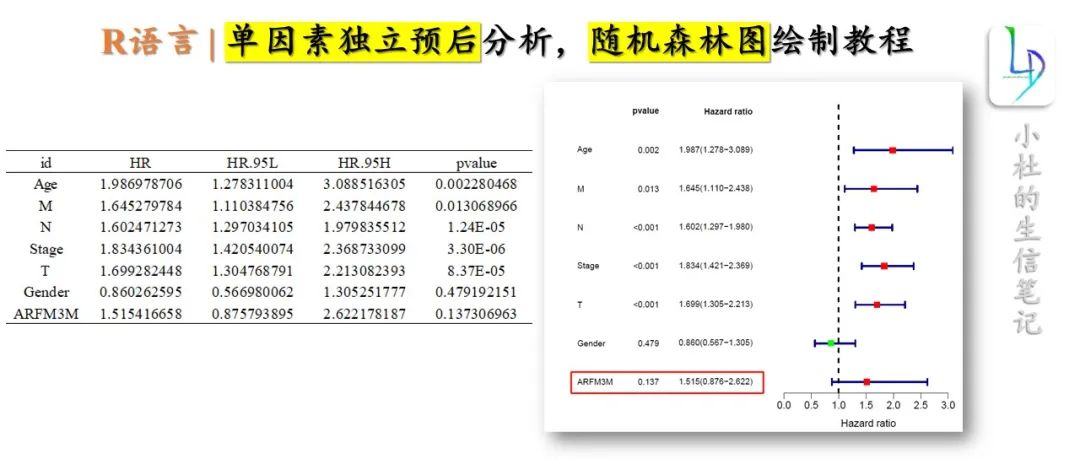
在医学研究中,为了研究临床病理特征与风险模型的预后情况,会将各种临床病理因素纳入风险模型,如年龄(Age),肿瘤分期(stage、T、 M、N)等,进行单因素Cox独立预后分析。并基于TCGA数据集,将目标基因表达状态和临床指标分别进行的单因素和多因素Cox回归分析,从而阐述目标基因与临床病理因素之间的关系。
今天的教程,我们先分享独立预后单因素Cox分析,并绘制随机森林图。
分析代码
- 加载R包
##'@加载对应R包
library(survey)
library(survival)
library(survminer)
- 导入数据
##'@导入数据
data_df <- read.csv("ARFM3M_input_data.csv", header = T, check.names = F, row.names = 1)
head(data_df)

3, 单因素独立预后分析
##'@单因素独立预后
rownames(data_df) <- data_df$id
head(data_df)
##
#data_df <- data_df[,-1]
data_df$Age = ifelse(data_df$Age >= 65,2,1)
##
data_df$futime <- as.numeric(data_df$futime)
data_df$fustat <- as.numeric(data_df$fustat)
#
data_df <- data_df[!data_df$futime == 0,]
##
class(data_df)
outTab <- data.frame()
for(i in colnames(data_df[,3:ncol(data_df)])){
cox <- coxph(Surv(futime, fustat) ~ data_df[,i], data = data_df)
coxSummary = summary(cox)
coxP=coxSummary$coefficients[,"Pr(>|z|)"]
outTab=rbind(outTab,
cbind(id=i,
HR=coxSummary$conf.int[,"exp(coef)"],
HR.95L=coxSummary$conf.int[,"lower .95"],
HR.95H=coxSummary$conf.int[,"upper .95"],
pvalue=coxSummary$coefficients[,"Pr(>|z|)"])
)
}
##'@单因素独立预后
rownames(data_df) <- data_df$id
head(data_df)
##
#data_df <- data_df[,-1]
data_df$Age = ifelse(data_df$Age >= 65,2,1)
##
data_df$futime <- as.numeric(data_df$futime)
data_df$fustat <- as.numeric(data_df$fustat)
#
data_df <- data_df[!data_df$futime == 0,]
##
class(data_df)
outTab <- data.frame()
for(i in colnames(data_df[,3:ncol(data_df)])){
cox <- coxph(Surv(futime, fustat) ~ data_df[,i], data = data_df)
coxSummary = summary(cox)
coxP=coxSummary$coefficients[,"Pr(>|z|)"]
outTab=rbind(outTab,
cbind(id=i,
HR=coxSummary$conf.int[,"exp(coef)"],
HR.95L=coxSummary$conf.int[,"lower .95"],
HR.95H=coxSummary$conf.int[,"upper .95"],
pvalue=coxSummary$coefficients[,"Pr(>|z|)"])
)
}
- 绘制随机森林图
####'@绘制图形单因素森林图
rt <- read.csv("unoCox_result.csv", header = T, row.names = 1)
gene <- rownames(rt)
hr <- sprintf("%.3f",rt$"HR")
hrLow <- sprintf("%.3f",rt$"HR.95L")
hrHigh <- sprintf("%.3f",rt$"HR.95H")
Hazard.ratio <- paste0(hr,"(",hrLow,"-",hrHigh,")")
pVal <- ifelse(rt$pvalue<0.001, "<0.001", sprintf("%.3f", rt$pvalue))
#
#输出图形
pdf(file="ATAD3A.unicox.forest.pdf", width = 7,height = 6)
n <- nrow(rt)
nRow <- n+1
ylim <- c(1,nRow)
layout(matrix(c(1,2),nc=2),width=c(3,2.5))
#绘制森林图左边的临床信息
xlim = c(0,3)
par(mar=c(4,2.5,2,1))
plot(1,xlim=xlim,ylim=ylim,type="n",axes=F,xlab="",ylab="")
text.cex=0.8
text(0,n:1,gene,adj=0,cex=text.cex)
text(1.5-0.5*0.2,n:1,pVal,adj=1,cex=text.cex);text(1.5-0.5*0.2,n+1,'pvalue',cex=text.cex,font=2,adj=1)
text(3,n:1,Hazard.ratio,adj=1,cex=text.cex);text(3,n+1,'Hazard ratio',cex=text.cex,font=2,adj=1,)
#绘制森林图
par(mar=c(4,1,2,1),mgp=c(2,0.5,0))
xlim = c(0,max(as.numeric(hrLow),as.numeric(hrHigh)))
plot(1,xlim=xlim,ylim=ylim,type="n",axes=F,ylab="",xaxs="i",xlab="Hazard ratio")
arrows(as.numeric(hrLow),n:1,as.numeric(hrHigh),n:1,angle=90,code=3,length=0.05,col="darkblue",lwd=2.5)
abline(v=1,col="black",lty=2,lwd=2)
boxcolor = ifelse(as.numeric(hr) > 1, 'red', 'green')
points(as.numeric(hr), n:1, pch = 15, col = boxcolor, cex=1.3)
axis(1)
dev.off()

> 在社群中可获得本期教程全部代码和示例数据
若我们的教程对你有所帮助,请点赞+收藏+转发,这是对我们最大的支持。
往期部分文章
「1. 最全WGCNA教程(替换数据即可出全部结果与图形)」
「2. 精美图形绘制教程」
「3. 转录组分析教程」
「4. 转录组下游分析」
「小杜的生信筆記」 ,主要发表或收录生物信息学教程,以及基于R分析和可视化(包括数据分析,图形绘制等);分享感兴趣的文献和学习资料!!