学习内容
LRT
尽管基因表达的静态测量很受欢迎,但生物过程的时程捕获对于反映其动态性质至关重要,特别是当模式复杂且不仅仅是上升或下降时。在处理此类数据时,似然比检验 (LRT) 特别有用。我们可以使用 LRT 来探索一系列时间点之间是否存在任何显著差异,并进一步评估样本类之间观察到的差异。
例如,假设我们有一个实验来观察随着时间的推移治疗对两种不同基因型小鼠的影响。我们可以为我们的“完整模型”使用一个设计公式,其中包括我们数据中的主要变异来源:genotype, treatment, time 和我们感兴趣的主要条件,即治疗效果随时间的差异(treatment:time)。
## 示例
full_model <- ~ genotype + treatment + time + treatment:time
要执行 LRT 测试,我们还需要提供一个简化模型,即没有 treatment:time 项的完整模型:
reduced_model <- ~ genotype + treatment + time
然后,我们可以使用以下代码运行 LRT:
dds <- DESeqDataSetFromMatrix(countData = raw_counts, colData = metadata, design = ~ genotype + treatment + time + treatment:time)
dds_lrt_time <- DESeq(dds, test="LRT", reduced = ~ genotype + treatment + time)
为了理解什么样的基因表达模式将被识别为差异表达,我们在下面有几个例子。在下图中,我们在 x 轴上有基因表达,在 y 轴上有时间。在此数据集中,每个时间点有两个样本,一个经过某种处理(红色),另一个未经过处理(蓝色)。
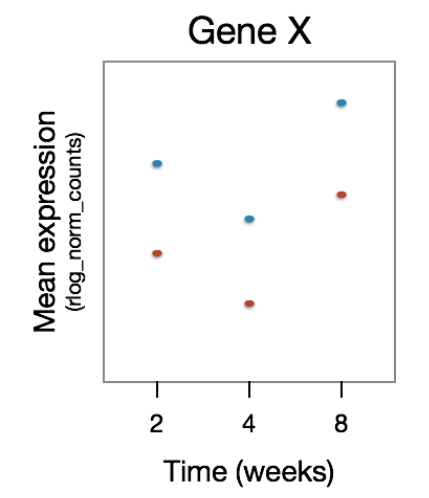
对于此图,我们描述了不会被识别为差异表达的基因类型。在这里,我们观察到 GeneX 在时间点之间有差异表达,但是治疗组之间的表达模式没有差异。
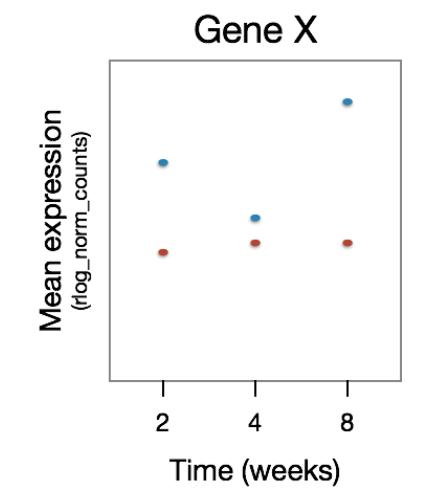
我们确实期望 LRT 返回的基因表达模式类型是那些随着时间的推移表现出治疗效果差异的模式。在下面的示例中,GeneX 显示了两个治疗组随时间变化的不同表达模式。
继续我们的示例数据集,在运行 LRT 之后,我们可以使用 padj < 0.05 的阈值来确定显着基因集。下一步是根据共享的表达模式将这些基因分类,我们可以使用 degPatterns() 来完成。在这里,您会注意到我们使用了 col 参数,因为我们有两个正在相互比较的组。
clusters <- degPatterns(cluster_rlog, metadata = meta, time="time", col="treatment")
根据数据中存在的共享表达谱类型,您可以提取与感兴趣的模式相关的基因组,并继续对每个感兴趣的基因组进行功能分析。 | ![]() 转录组
426 人阅读
|
0 人回复
|
2024-06-28
转录组
426 人阅读
|
0 人回复
|
2024-06-28