1. 基本概念
CNVs(Copy Number Variantion): 即拷贝数变异,与生长发育迟缓、行为言语障碍以及神经发育迟缓、自闭症谱系障碍等疾病相关。
ROH(Region Of Homozygosity): 纯合性区域,指在拷贝数正常状态下,该区域内基因组序列表现为纯合,导致ROH的原因可能为UPD。
LOH(Loss Of Heterozygosity): 杂合性缺失,表现为该区域内不存在杂合的状态。
AOH(Absence Of Heterozygosity): 杂合性不存在,和LOH一样。
UPD:单亲二体,指父或母一方的染色体片段被另一方的同源部分取代,或某一个体的两条同源染色体都来自同一亲本,主要发生在胚胎期,常表现为CPM。普通人群中,父源UPD与母源UPD比例为1:3, 且UPD常出现在16号、4号、22号、1号、21号和X染色体。UPD在高龄孕妇中更容易发生;UPD是产前发育异常、儿童智力底下和发育迟缓等疾病中重要的原因(新生儿UPD发生率约1/2000)。
单亲异二体(heterodisomy, UPhD):指两条染色体来自同一亲本的两条同源染色体。
单亲同二体(isodisomy, UPiD):指两条染色体来自同一亲本的同一染色体,会导致隐性遗传基因突变的激活。
复合型单亲二体(mix-UPD):部分表现为单亲同二体,部分表现为单亲异二体。
片段性单亲二体(segmental UPD, seg-UPD):指染色体的一部分片段表现为UPD。
CPM(Confined Placental Mosaicism): 限制性胎盘嵌合 ,指异常细胞系几乎只存在于胎盘,而不存在于羊膜细胞或其他胎儿组织中,即染色体嵌合异常仅存在于胎盘而胎儿染色体正常的现象。
IBD(Identity-By Descent):血缘同源,亲属个体基因序列由某一共同祖先基因拷贝而来,异常同源染色体分别来自父母,基因序列完全相同,导致出现纯合区域,大多数由于近亲结婚导致,导致ROH的原因可也能为IBD。
VAF(Variant Allele Frequency):变异等位基因频率,即基因组某个位点支持alternate/mutant allele的reads覆盖深度占这个位点总reads覆盖深度的比例。VAF计算公式:VAF=AD/DP, 其中AD代表Allele Depth, DP代表Total Depth。
在二倍体胚系分型中,杂合位点的VAF在高测序深度下比例应接近0.5, 表示该位点allele均存在一个拷贝;当VAF比例为0.25 / 0.75时,表示基因组能存在另一个拷贝。肿瘤组织(或ctDNA)变异检测结果会包含正常的allele和突变的allele,通过计算VAF可推断肿瘤的异质性和肿瘤纯度(tumor purity),并且VAF的高低可能会影响癌症的预后。
MAF(Minor Allele Frequency):次等位基因频率,在群体遗传学中表示群体中丰度次高(第二高)的allele的频率,用来区分某个allele是多态性还是稀有变异。
肿瘤纯度(tumor purity):指的是样本中肿瘤细胞占所有细胞的比例。肿瘤组织取样混入正常细胞会对后续分析产生影响,因此生信分析中需要借助肿瘤纯度对分析进行矫正。
2. CNV-seq简介
CNV-seq为拷贝数变异测序,是一种全基因组测序,可以检测基因组23对染色体的非整倍体及大于100kb的CNVs,因此可准确定位到致病CNVs。CNV-seq一般针对的是流产物或全血,可运用于不孕不育和反复流产原因排查、产前超声结构异常检测、无创产前检测、羊水穿刺检测、复杂疾病和严重遗传病致病因素排查(诸如先天性心脏病、威廉姆斯综合征、癫痫、唐氏综合征等)、辅助生殖、法医检测、肿瘤早筛、嵌合体排查等。
CNV-seq 数据分析:
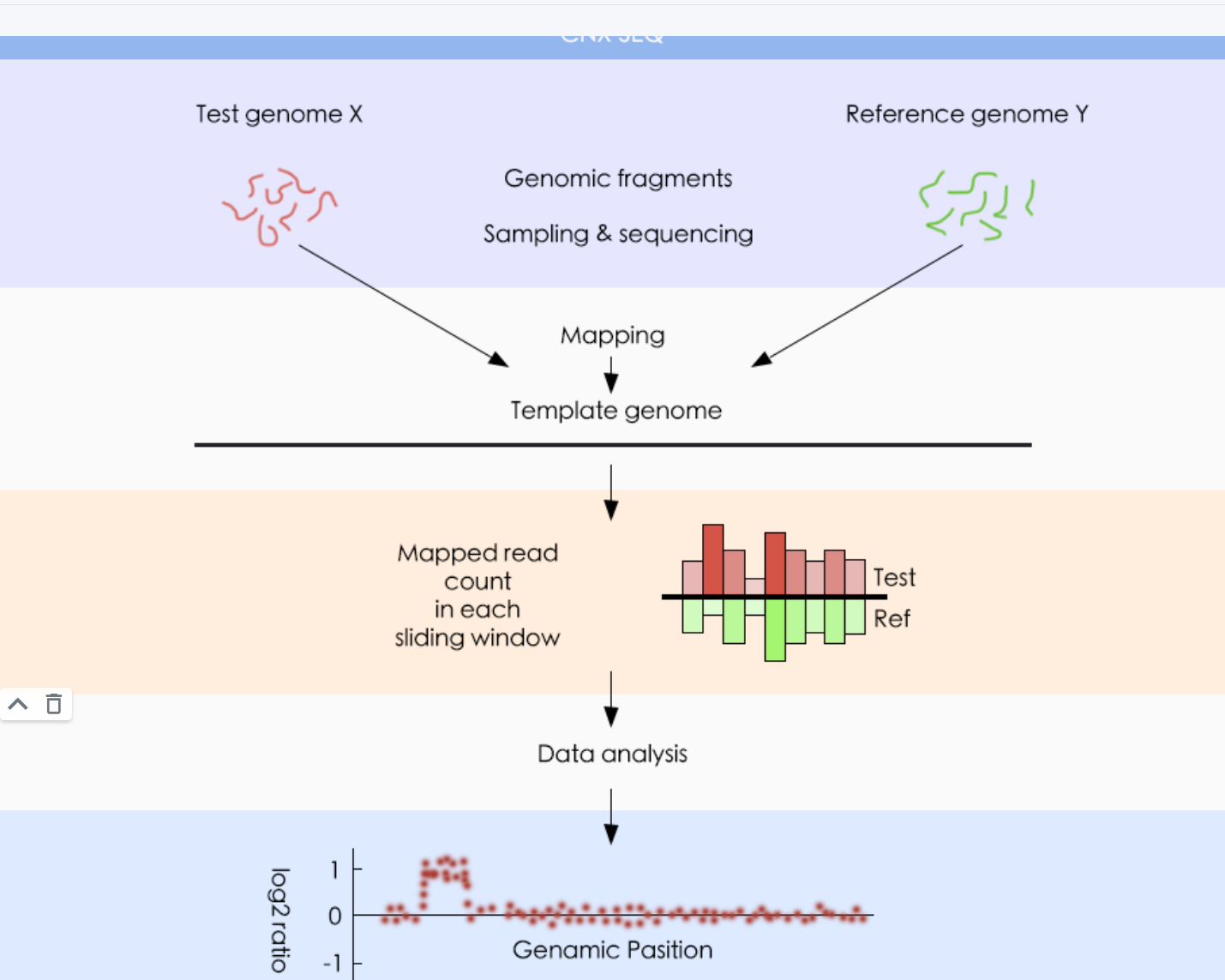
2.1 低深度CNV-seq
低深度全基因组测序,低深度CNV-seq检测项目一般测序深度大多在0.05x至1X,一般分辨率可到100kb,无法获取大多数SNPs位点的杂合性和等位基因频率(AF),因此CNV-seq无法检出包含单亲二体(UPD)在内的杂合性缺失(LOH。
2.2 中深度CNV-seq
中深度CNV-seq检测项目一般测序深度大多在3X,相比于低深度CNV-seq,CNVs检出率和分辨率有大幅提升,在变异检测时选取人群频率高的位点,使用滑窗对位点的VAF进行平滑处理能进行ROH的检测。
非唯一比对区域、重复区域、或GC异常区域,无论是CMA还是低深度/中深度CNV-seq都是检测的灰区(或称为盲区。)
3. CNVs的评级
对于CNV-seq检测到CNVs:
(1)先评估CNVs区域涉及的编码蛋白的基因,是否涉及已明确的疾病;
(2)DGV数据库查看该CNVs在正常人群中是否存在;
(3)ClinGen数据库查看该CNVs是否涉及剂量敏感区域;
(4)Decipher数据库是否能检索到该CNVs的病例/案例;
(5)PubMed是否能检索到与该CNVs相关的已发表的文献报道。