1.Bcftools 从VCF/BCF文件中提取信息
bcftools query命令可用于提取任何VCF字段。
# 查看vcf文件包含样本名称
bcftools query -l sample.vcf
# 查看vcf文件包含样本数量
bcftools query -l sample.vcf| wc -l
# 打印POS列信息, head显示前10列
bcftools query -f '%POS\n' sample.vcf|head
# 打印CHROM POS REF ALT 4列信息
bcftools query -f '%CHROM %POS %REF %ALT\n' sample.vcf|head
# chr1 69270 A G
2. Bcftools 提取每个位点的等位基因频率
# 提取每个位点的等位基因频率
bcftools query -f '%CHROM %POS %AF\n' sample.vcf |head
# chr1 69270 1
# 如果AF注释不存在,但AN和AC存在,可计算频率
bcftools query -f '%CHROM %POS %AN %AC{0}\n' sample.vcf | \
awk '{printf "%s %s %f\n",$1,$2,$4/$3}' |head
# 1 13380 0.000077
3. Bcftools 提取每个样本标签
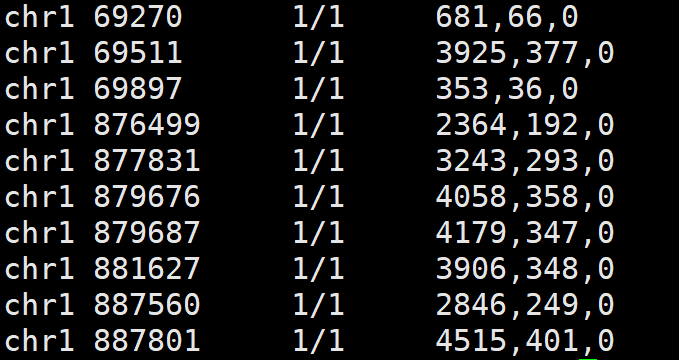
# 提取CHROM POS 基因型和PL列信息
bcftools query -f '%CHROM %POS[\t%GT\t%PL]\n' sample.vcf |head
4. Bcftools 按固定列过滤
固定列如QUAL、FILTER、INFO可以直接过滤。
# 使用-e 'FILTER="."'表达式排除不是FILTER的位点
bcftools query -e 'FILTER="."' \
-f '%CHROM %POS %FILTER\n' sample.vcf |head
# 1 3000150 PASS
# 1 3000151 LowQual
4. Bcftools 按质量和深度过滤
bcftools query -i 'QUAL>20 && DP>10' \
-f '%CHROM %POS %QUAL %DP\n' sample.vcf | head
# 1 14930 31.2757 13
# 1 17538 37.9458 12
5. Bcftools mpileup生成可能基因型文件
BAM文件作为输出,输出可能基因型的BCF文件。
bcftools mpileup \
--max-depth 10000 \
--threads 4 \
-f reference.fasta \
-o genotype.bcf \
sample.bam
参数说明:
--max-depth或-d:为比对中的每个位置设置每个输入文件的读数。在本例中,设置为10000;
--threads:设置要使用的处理器/线程的数量(n);
--fasta-ref或-f:用于选择用于比对的faidx索引的FASTA核苷酸参考文件(reference.fasta);
--output 或-o:输出文件(genotype.bcf);
最后一个参数:输入BAM比对文件(sample.bam), 可以提供多个输入文件。
6. Bcftools call生成SNP/indel变异文件
bcftools call可用于从BCF文件call SNP/indel变异。
bcftools call \
-O v \
--threads 4 \
-vc --ploidy 2 \
-p 0.05 \
-o variants.unfiltered.vcf \
genotype.bcf
参数说明:
--output-type或-O:用于选择输出格式。在本例中,v表示VCF;b表示生成BCF。
--threads:设置要使用的处理器/线程的数量(n)。
-vc:指定输出仅包含变体。
--ploidy:指定程序的倍性(人类为二倍体)。
--pval-threshold或-p:用于设置变异位点的p值阈值(0.05)。
--output 或-o:输出文件(variants.unfiltered.bcf)。
最后一个参数:输入BCF文件(genotype_likelihoods.bcf)。
7. Bcftools filter变异文件
bcftools filter可用于从BCF文件中过滤变异。
bcftools filter \
--threads n \
-i '%QUAL>=20' \
-O v \
-o variants.filtered.vcf \
variants.unfiltered.vcf
参数说明:
--threads:设置要使用的处理器/线程的数量(n)。
--include或-i:过滤表达式, %QUAL>=20表示筛选出质量大于或等于20位点。
--output-type或-O:输出格式,,v代表VCF。
--output 或-o:输出文件(variants.filtered.vcf)。
最后一个参数:输入BCF文件(variants.unfiltered.vcf)。
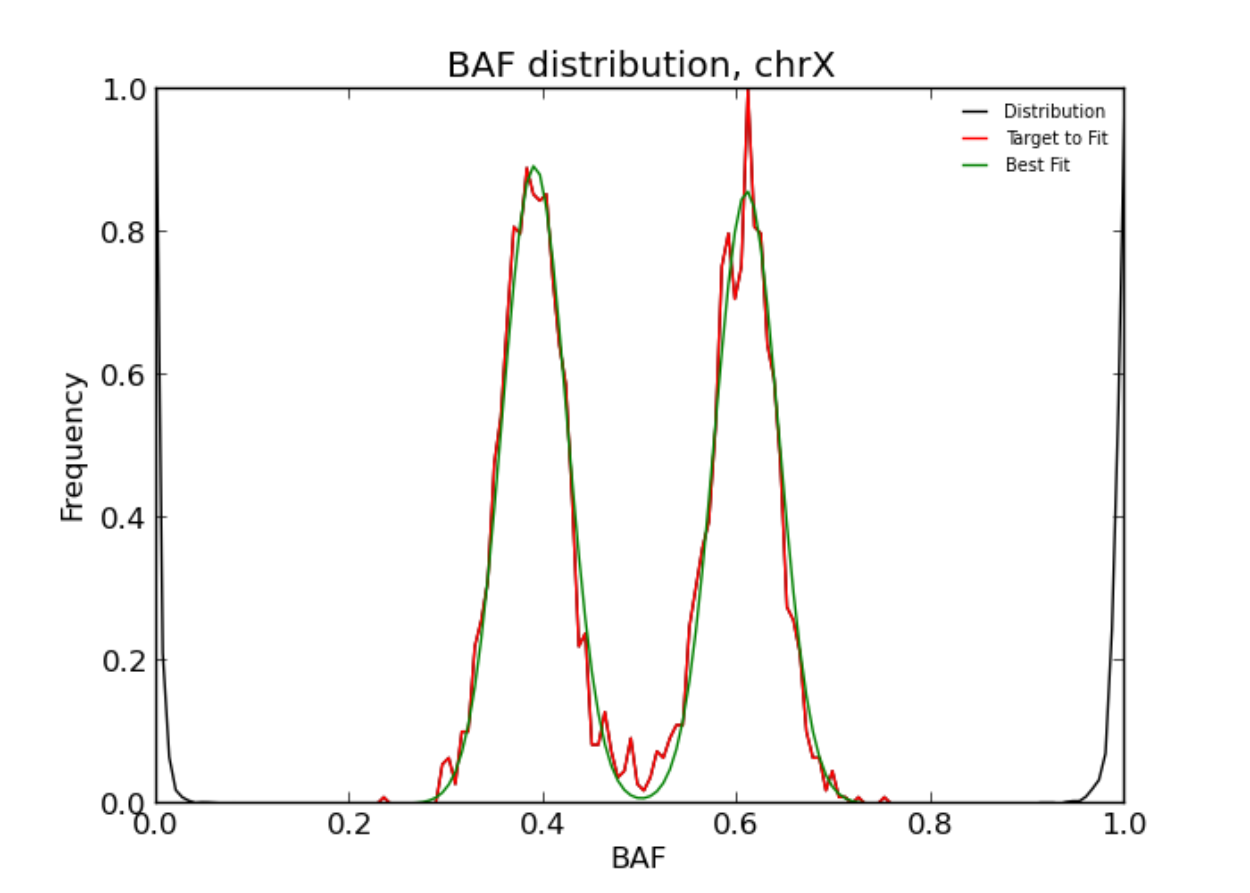
8. 检测非整倍性和污染
检测影响整个染色体的SV,如非整倍性或污染,可以直接从BAF值的总体分布中识别。
bcftools polysomy -v -o outdir/ sample.vcf
# 对于二倍体,获取第三列值缺失1.0和重复3.0的行
cat outdir/dist.dat | awk '$1=="CN" && $3!=2.0'
# matplotlib绘图
python outdir/dist.py
下图输出的结果图中,有三个X染色体拷贝。
9. 23andMe转换为VCF
转换只考虑SNP而忽略缺失和插入。
# 原始的23andMe结果示例
# 四列分别为:标记ID,染色体名称,位置和基因型
#rs6139074 20 63244 AA
#rs1418258 20 63799 CC
#rs6086616 20 68749 TT
#rs6039403 20 69094 AG
bcftools convert \
--tsv2vcf sample.23andMe.gz \
-f reference.fa \
-s sample_name \
-Ov \
-o sample.vcf
# Rows total: 612647
# Rows skipped: 4751 (跳过基因型)
# Missing GTs: 20525 (缺失基因型)
# Hom RR: 318339
# Het RA: 165598
# Hom AA: 103420
# Het AA: 14
# Het AA数量应该比较小,因为大量的杂合等位基因(Het AA)表明输入等位基因不在正链上。
10. 合并到多个样本的VCF文件
bcftools index sample1.vcf
bcftools index sample2.vcf
# 合并为merge.vcf
bcftools merge -Ov -o merge.vcf sample1.vcf sample2.vcf
11. VCF文件添加注释信息
输入VCF/BCF文件、fasta格式的参考基因组和可从Ensembl网站下载的GFF 3文件,输出包含注释信息的VCF文件,目前,仅支持Ensembl GFF 3文件。
bcftools csq \
-f reference.fa \
-g Homo_sapiens.GRCh37.82.gff3.gz \
sample.vcf \
-Ov \
-o output.vcf
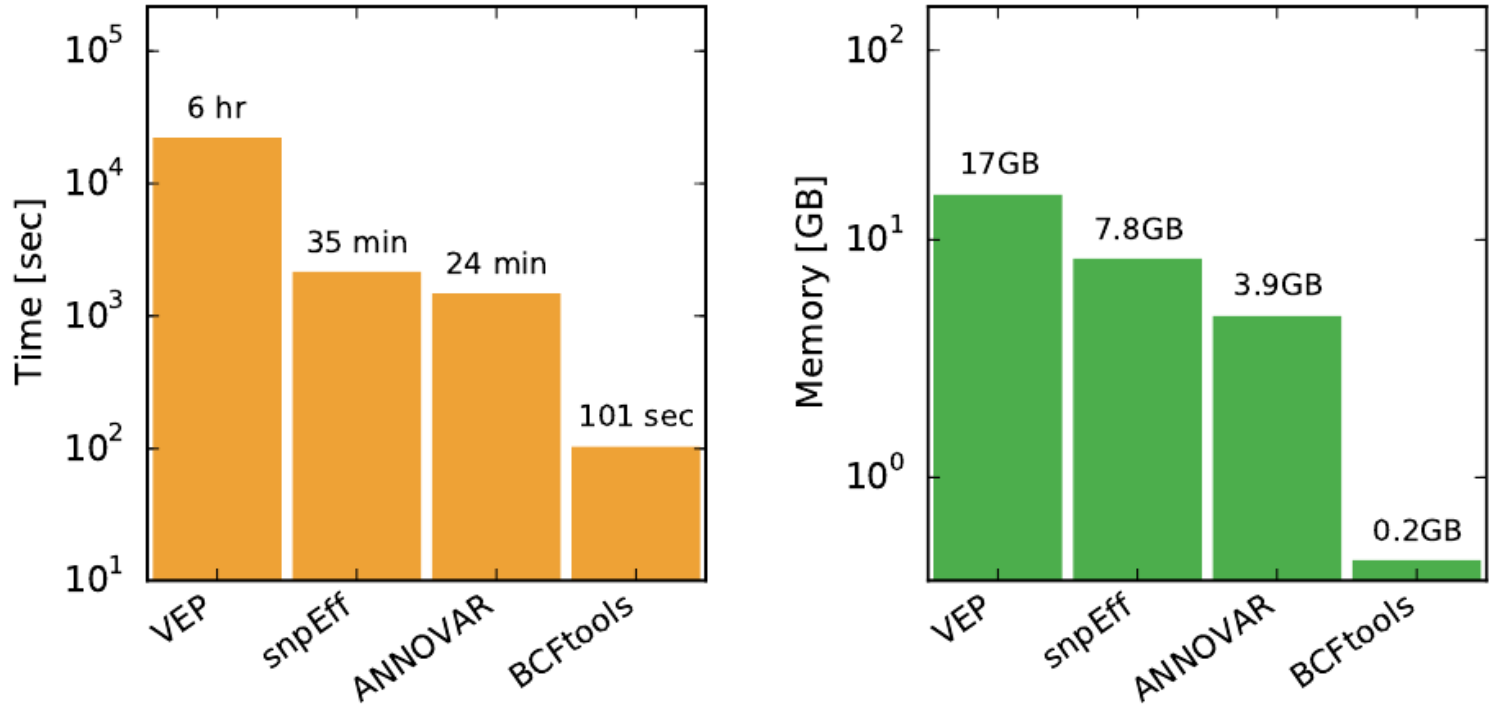
bcftools与三款主流变异注释软件速度和内存消耗比较。