今天推文的内容可以在阿里云服务器上完成(配置2核2G 40G存储)购买费用是99元一年,如果是完全新用户,现在是82一年。可以通过以下链接购买
https://www.aliyun.com/daily-act/ecs/activity_selection?source=5176.11533457&userCode=3enjgk6n
如果通过这个链接购买了云服务器,需要本期视频的示例数据,可以添加我的微信 mingyan24
本篇推文中用到的流程是参考论文
Super-pangenome analyses highlight genomic diversity and structural variation across wild and cultivated tomato species
https://www.nature.com/articles/s41588-023-01340-y
需要用到的软件是
bcftools (这个软件用conda或者源码编译在云服务器上都比较麻烦,如果自己没有搞定的话可以添加我的微信帮忙解决)
vcftools
plink
emmax https://csg.sph.umich.edu//kang/emmax/download/index.html
the Genetic type 1 Error Calculator https://pmglab.top/gec/#/download
R语言
R包 qqman
做GWAS分析需要准备的输入数据
我准备的表型数据格式如下
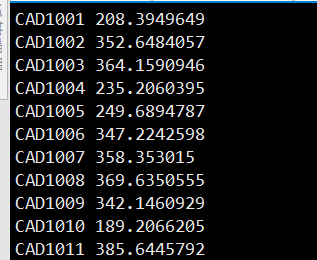
两列,第一列是样本名,第二列是表型值 制表符分隔
EMMAX这个软件要求的表型输入格式是3列,前两列都是样本名,第三列是表型值,运用awk命令重新生成一个表型文件
cat phenotype.txt | awk '{print $1"\t"$1"\t"$2}' > pheno_emmax.txt
vcf文件中的样本顺序需要和表型文件中的样本顺序一致,调整一下vcf文件中的样本顺序
cat phenotype.txt | awk '{print $1}' > sample.order
bcftools reheader -s sample.order smoove.filtered.impute.vcf.gz | bcftools view -m2 -M2 -O v -o output.vcf
-m2 -M2 这两个参数是只输出2等位的位点
对vcf文件进行过滤
标准是最小等位基因频率和缺失率
(SNP-based GWAS was perfored using SNPs with minor allele frequency > 0.01 and missing call rate < 0.1.)
vcftools --vcf output.vcf --max-missing 0.9 --maf 0.05 --recode --recode-INFO-all --out output.filter
计算主成分
Population structure was calculated by principal component analysis in PLINK (v.1.9.0b4.6)91 using 437,028 SNPs showing less linkage disequilibrium, which was extracted using PLINK with parameters‘–indep-pairwise 50 5 0.1 (windows, step, r2)’.
首先是根据 LD水平对对数据集进行过滤
plink --vcf output.filter.recode.vcf --recode12 --allow-extra-chr --allow-no-sex --out smoove
plink --allow-extra-chr --file smoove --indep-pairwise 50 5 0.1 --recode vcf-iid --out smoove.LDpruned
plink --allow-extra-chr --file smoove --recode vcf-iid --extract smoove.LDpruned.prune.in --out smoove.LDpruned
这一步会生成一个smoove.LDpruned.vcf 这里只有500 多个位点了原来是8000多个位点
用过滤后的数据集计算主成分
plink --allow-extra-chr --vcf smoove.LDpruned.vcf --make-bed --out LDpruned.bfile
plink --bfile LDpruned.bfile --allow-extra-chr --allow-no-sex --pca 10 --out PCA
cat PCA.eigenvec | awk '{print $1,$2,"1",$3,$4,$5,$6,$7}' > pca.covariates
## 选用前5个主成分作为协变量
emmax计算情缘关系矩阵
plink --file smoove --allow-extra-chr --recode 12 --transpose --out smoove_
~/biotools/emmax/emmax-kin-intel64 -v -d 10 smoove_ -M 1
这里 -M参数指定需要用到的最大内存是 1G
这里输出的亲缘关系矩阵全是nan
这里应该是染色体编号不标准的原因,这个示例vcf文件的染色体编号是 Gm01 Gm02,把smoove_.tped文件里的这个文件里的Gm0 Gm替换成空试试,染色体号完全是数字
sed -i 's/Gm0//g' smoove_.tped
sed -i 's/Gm//g' smoove_.tped
~/biotools/emmax/emmax-kin-intel64 -v -d 10 smoove_ -M 1
这样就有结果了
emmax主成分分析
~/biotools/emmax/emmax-intel64 -v -d 10 -t smoove_ -p pheno_emmax.txt -k smoove_.aBN.kinf -c pca.covariates -o output
output.ps 这个文件的最后一列是 gwas分析的p值
把染色体编号 位点 和p值合并到一起
cat smoove_.map | paste - output.ps | awk '{print $2,$1,$4,$8}' | sed '1i\SNP CHR BP P' | sed 's/ /\t/g' > gwas.output
计算显著性阈值
cp output.vcf output.backup.vcf
sed -i 's/Gm0//g' output.backup.vcf
sed -i 's/Gm//g' output.backup.vcf
plink --vcf output.backup.vcf --allow-extra-chr --make-bed --out gec.input
java -jar -Xmx1g ~/biotools/gec/gec.jar --effect-number --plink-binary gec.input --genome --out gec.output
输出文件 gec.output.sum suggestive_P_value 2.21E-4
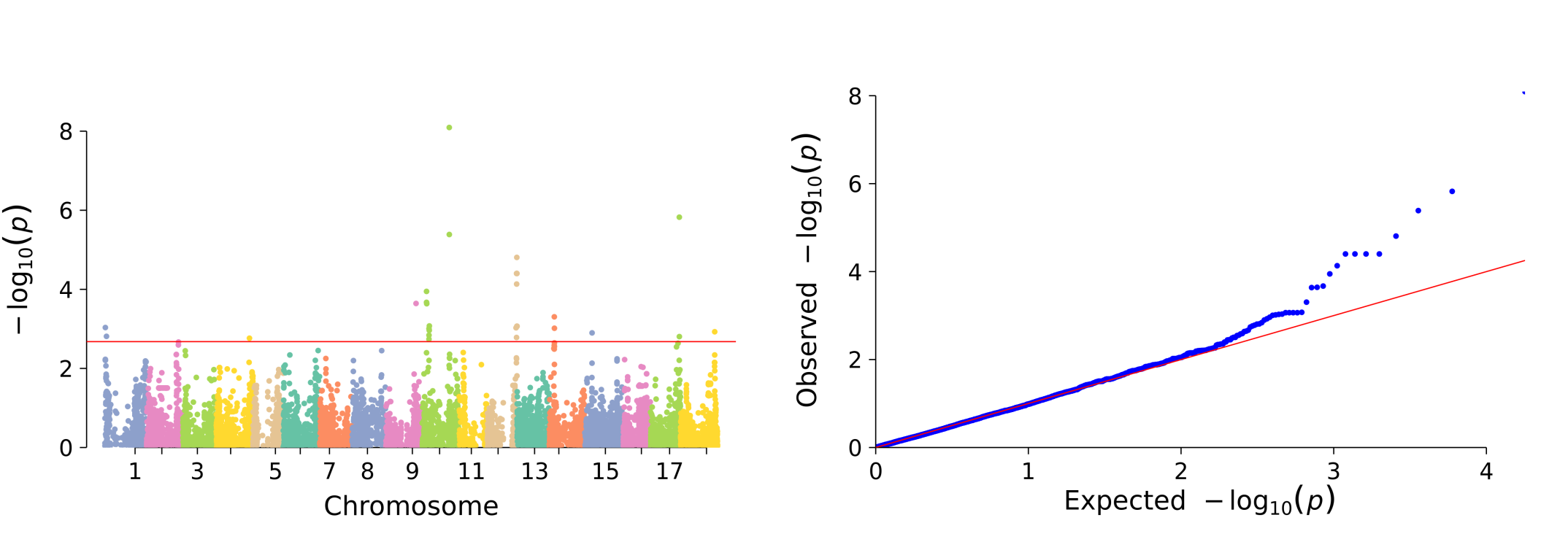
画曼哈顿图
sed -i 's/Gm0//g' gwas.output
sed -i 's/Gm//g' gwas.output
Rscript manhattan_qq.R gwas.output manhattanPlot.png 0.0021
欢迎大家关注我的公众号
小明的数据分析笔记本
小明的数据分析笔记本 公众号 主要分享:1、R语言和python做数据分析和数据可视化的简单小例子;2、园艺植物相关转录组学、基因组学、群体遗传学文献阅读笔记;3、生物信息学入门学习资料及自己的学习笔记!
![]() 遗传学
1631 人阅读
|
0 人回复
|
2024-06-04
遗传学
1631 人阅读
|
0 人回复
|
2024-06-04