代码链接
https://github.com/eriqande/make-ancestral-fasta-snakeflow/tree/main
这个是一个snakemake的流程
这里推断祖先染色体就是选择一个近缘种的基因组序列,自己的基因组作为参考,近缘种的基因组作为查询序列与参考基因组进行比对,将snp位点的数据替换成近缘种的碱基序列
这里用两个拟南芥的基因组做测试(每个基因组里只用了一条染色体,两个染色体id不能是一样的)
比对代码
这里换成自己的物种数据具体用什么参数还需要仔细看看帮助文档
lastz an1.chr1.fa c24.chr1.fa --notransition --step=20 --inner=1000 --identity=90 --gapped --ambiguous=iupac --format=maf --chain > output.maf
对maf格式进行简化
single_cov2 output.maf > output01.maf
maf格式转换为fasta
maf2fasta an1.chr1.fa output01.maf fasta > output01.maf.fna
输出祖先染色体序列
Rscript condense_and_summarise_fastas.R output01.maf.fna output02.fna a.txt b.txt an1chr1
第一个参数是输入数据
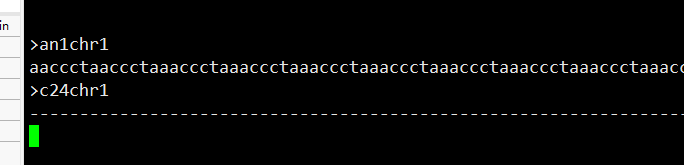 第二个参数是输出的结果
第二个参数是输出的结果
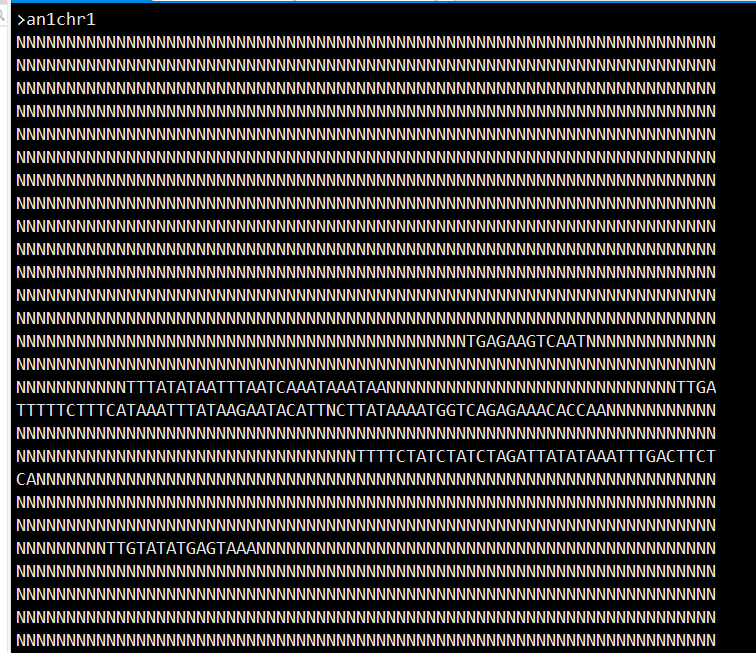
第三四个参数没太搞明白是啥
第五个参数是参考基因组的染色体id | ![]() 基因组
224 人阅读
|
0 人回复
|
2024-07-26
基因组
224 人阅读
|
0 人回复
|
2024-07-26