基因组之全局互作热图可视化
## 引言**PlotHiC** 是一个专为 Hi-C 数据可视化分析而设计的 Python 包。Hi-C 技术是一种能够检测染色体三维结构的实验方法,它能揭示 DNA 在细胞核内的三维组织结构。为了更好地展示和解释这些复杂的数据,(https://github.com/Jwindler/PlotHiC "Source") 可以帮助用户方便地绘制Hi-C 数据的热图。
- 优势
- 仅使用 `.hic`文件,无需 `merged_nodups.txt`,仅5秒即可出图
- 可自定义染色体名称
- 无需 `assembly`文件
- 更新
- 如果有新的需求或者问题请 (https://github.com/Jwindler/PlotHiC/issues/new)
## 安装
需要提前安装好 `python>3.10`
```sh
# pip install
pip install plothic
```
## 使用
### 输入文件
1. `.hic` : 该文件来自3d-dna,您需要选择最终的hic文件(已进行错误调整并确定染色体边界)。
2. `chr.txt` : 该文件用于在热图标记染色体的边界线和名称。第一列是染色体的名称,第二列是染色体的长度(这个长度是Juicebox中hi件的长度,可以从Juicebox手动确定)。
```sh
# name length
Chr1 24800000
Chr2 44380000
Chr3 63338000
Chr4 81187000
Chr5 97650000
```
### 示例
```sh
plothic -hic test.hic -chr chr.txt -r 100000
# -hic > .hic file
# -chr > chromosome length (in .hic file)
# -r > resolution to visualization
```
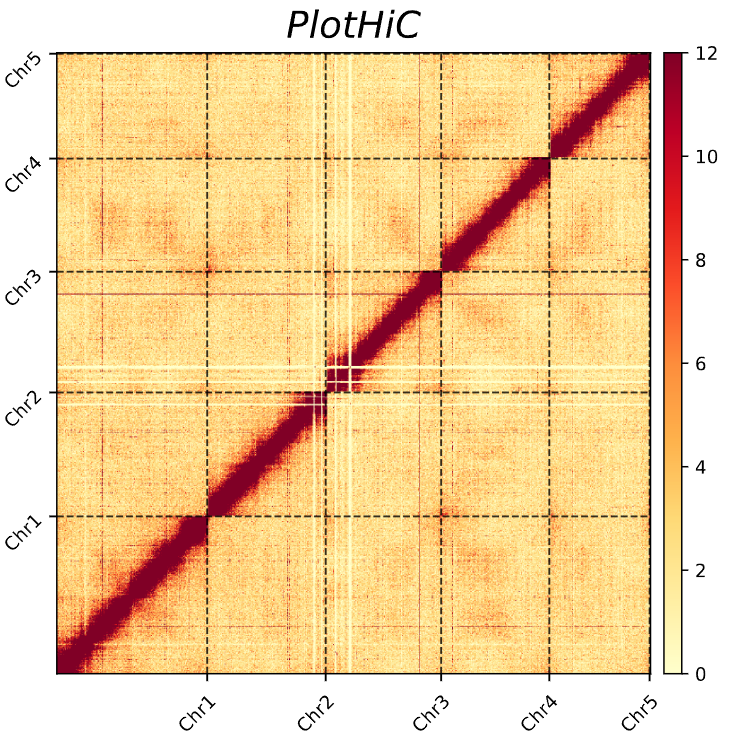
- 输出结果
页:
[1]